ライフサイエンスの分野では、計測技術の発展により、顕微鏡などを使って分子や細胞、組織、個体などの動体を生きたまま観察した計測データが得られつつあります。例えば、線虫の細胞分裂を顕微鏡で深さ方向や時間方向に連続的に撮影することで、高次元な画像データを得ることができます。このような画像データから、計算機で画像処理することで細胞核や胚の領域を特定し、その座標データを抽出することができます。以下のmp4動画(1.8MB)はその一例です。可視化や定量的な解析が可能になります。
そこで本研究室では、マウスや線虫、大腸菌などのさまざまな生物の計測データに対して、画像認識、異常検知、因果推論の3つのテーマに分かれて取り組んでいます。研究成果の一覧をこちらに、過去の卒業論文や修士論文のタイトルの一覧をこちらに示します。
画像認識
マウスの脳や線虫の胚を撮影した顕微鏡画像データを例に、画像内に撮影された物体がどこにあるかを推定するための「画像セグメンテーション」という技術の研究開発に取り組んでいます。
画像セグメンテーションは、画像内の対象物体と背景などを領域ごとに分ける手法です。人工知能や機械学習の手法を利用します。具体的には、ニューラルネットワークに画像とセグメンテーションの正解情報を与えることで、物体の位置を推定します。この学習は「教師あり学習」と呼ばれ、バイオメディカルの分野で得られる小規模な画像に効果的です。例えば、U-net と呼ばれる有名なニューラルネットワークのモデルは、バイオ医療画像のセグメンテーションに適しており、比較的小規模な画像データの学習でも、高い精度を実現します。

(左) 微分干渉顕微鏡画像と、(右) 細胞核の領域の正解データの例
しかし、撮影条件を微妙に変えて撮影した画像に対して高精度なセグメンテーションを達成するには、正解データを準備する必要があります。正解データの準備には高い専門知識を要するため、このような問題解決の方法は、現実的ではない場合があります。
そこで「教師なしドメイン適応」と呼ばれる学習手法を試みています。例えば、公共の画像データベースなどに公開されている大量の画像データで、ニューラルネットワークのモデルのパラメータを事前に学習します。そして、「敵対的に学習」として、撮影条件の異なる新しい画像データに対しても画像セグメンテーションができ、かつ、元の画像データのセグメンテーション結果と新しい画像のセグメンテーション結果を区別するよう学習する方法が有効です。さらに、深層学習の学習に、画像の時間的な変化や深さ方向の変化といった、生物学的な情報を活用する方法にも取り組んでいます。これにより、動的なプロセスや立体的な情報を考慮することが可能になります。
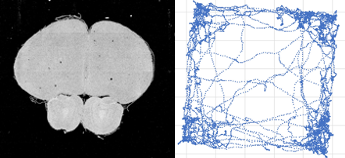
マウスの脳画像(下図左)と行動(右)データを統合的に解析することで、脳の神経細胞の誕生時期や位置、そして、そうした神経細胞の活性化や抑制が、マウスの行動にどのような影響を与えるかを明らかにすることを目指しています。これにより、脳と行動の複雑な関係を理解し、さらには、神経疾患や行動障害のメカニズムについての洞察を得ることを期待しています。
異常検知
正常な線虫の胚がどのように細胞分裂が進むかを顕微鏡で観察した「正常データ」と、線虫の1遺伝子の機能を著しく低下させた線虫の胚を撮影した「異常データ」で、どのような違いがあるかを計算機で自動的に検知し、定量的に比較することは、遺伝子の機能を推定する上で重要です。そこで、異常検知に関して、以下の2種類のアプローチに取り組んでいます。
1つめのアプローチは、深層学習の技術を用いた異常検知の研究です。異常データのすべてのバリエーションを把握し教師あり学習することは難しいため、正常データを AutoEncoder などニューラルネットワークのモデルで教師なし学習するアプローチが一般的です。正常データを学習したモデルに異常データを与え、再現できない部分を異常部位として検出します。しかし、AutoEncoder は、汎化性能が高すぎる性質があり、生物画像の異常検知に用いると、異常データも再現してしまい異常が検出できない問題があります。そのため、AutoEncoderの派生モデルであるMenAEの利用をはじめ、画像中の着目したい部分に注目したり、細胞の形状の異常や時間の異常を区別するモデルの構築に取り組んでいます。
2つめは、確率モデルの技術である隠れマルコフモデル(HMM)などを使用した異常検知の研究です。音声認識で用いられてきた技法です。時系列データをモデルに与え時系列データのパターンを認識し、異常検知をするために、HDP-HSMMと呼ばれるモデルの応用を試みています。こちらでは画像を直接与えるよりも、高い精度で異常検知することを目指します。
因果推論
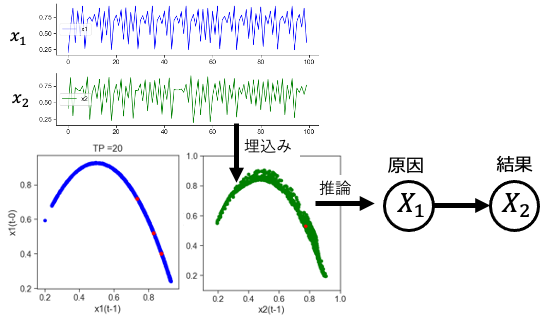
顕微鏡画像データを画像認識したり、生体内の代謝物を計測することで、さまざまな時系列データを得ることができます。そこで、時系列データ間で、原因と結果の関係があるかや、因果関係の強さ、方向などの推定する時系列因果推論の技術の研究しています。従来の時系列解析は線形な性質のデータ(平均や分散、その分布がどの時間でも変化しない)を対象にしていました。しかし、本研究室では、生物データに対する時系列因果推論を実現するために、非線形な性質の時系列データに注目し、時系列長が短く、ノイズを多く含むようなデータに対して頑強な手法を提案を目指しています。
時系列因果推論の従来手法には、線形な時系列に適した Granger 因果性テストや、非線形な時系列に適した Convergent Cross Mapping (CCM) という手法があります。かなり独自色の強いテーマで、立命館大学情報理工学部では唯一、国内でも本テーマに取り組んでいる研究室は限られます。
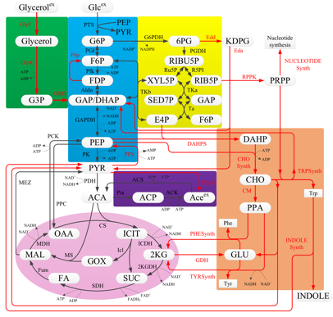
なお、因果推論の研究の過程で、大腸菌の細胞増殖と糖を代謝を再現する数理モデルの構築や、モデルのパラメータ最適化のアルゴリズムの研究も行っています。下図はグルコースをATPなどに分解するエネルギー代謝を、常微分方程式で記述した数理モデルの全体像を表しています。こうした数理モデルの構築とパラメータの最適化、シミュレーションにより、細胞の分裂や増殖における重要な要因や、因果推論の手法を組み合わせることで、生育環境の変化が生体内に及ぼす影響を推定しようとしています。